Arbeitsgebiete
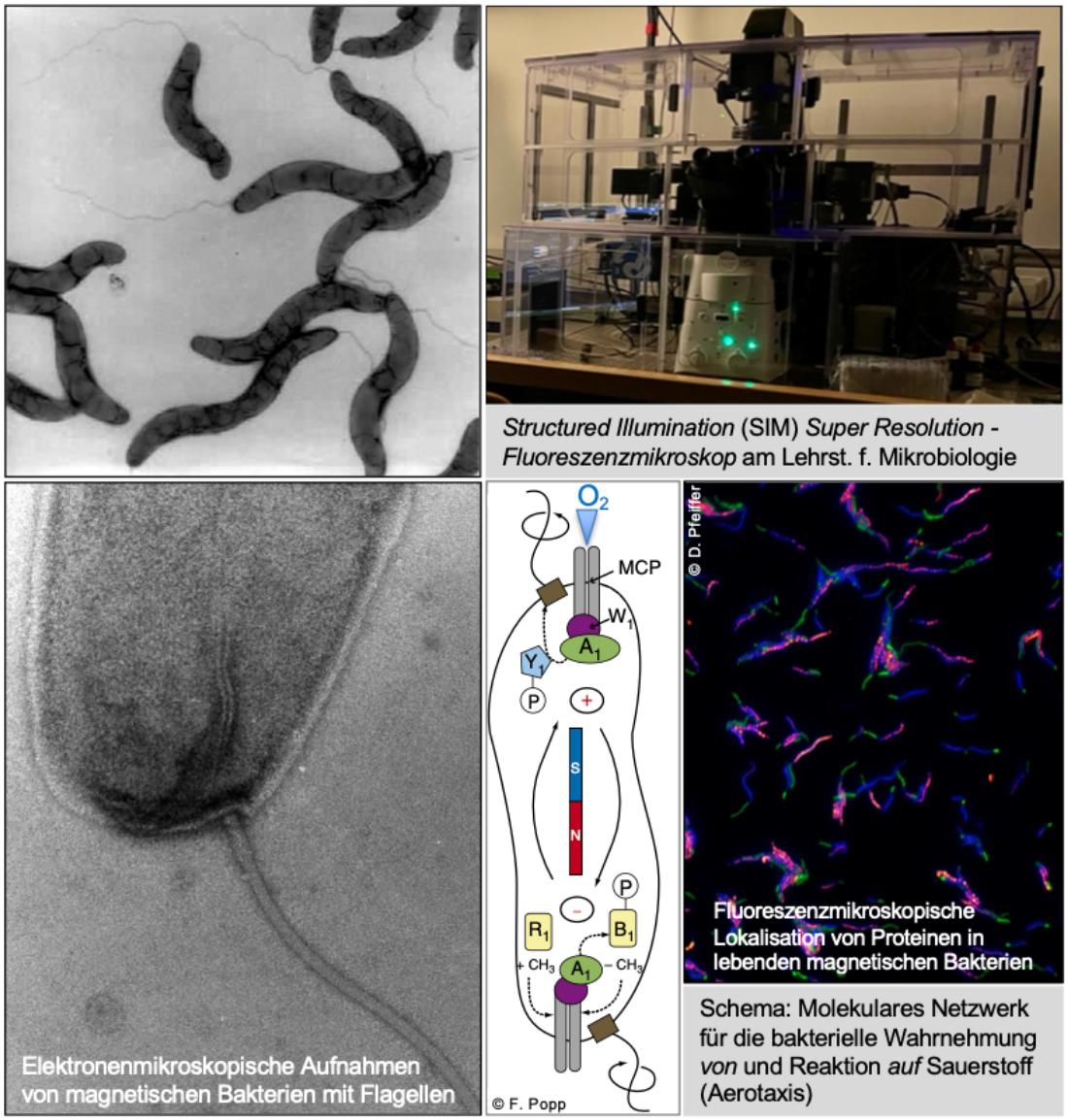
Molekulare Mechanismen der bakteriellen Magneto-Aerotaxis
Magnetosomen dienen als zelluläre "Sensoren" für die passive Ausrichtung der Bakterien im Erdmagnetfeld. Die aktive Schwimmbewegung entlang der magnetischen Feldlinien mit Hilfe von rotierenden Flagellen im Zusammenwirken mit einer empfindlichen Aerotaxis ermöglicht magnetotaktischen Bakterien das Aufsuchen von Regionen mit optimaler Sauerstoff-Konzentration im Sediment von Gewässern. Wir untersuchen die zugrunde liegenden molekularen Mechanismen sowie die Zellbiologie dieser Magneto-Aerotaxis.
Weitere Informationen:
Pfeiffer, D., M. Toro-Nahuelpan, R.P. Awal, F.D. Müller, M. Bramkamp, J.M. Plitzko, and D. Schüler. 2020. A bacterial cytolinker couples positioning of magnetic organelles to cell shape control. Proc. Natl. Acad. Sci. USA, doi: 10.1073/pnas.2014659117.
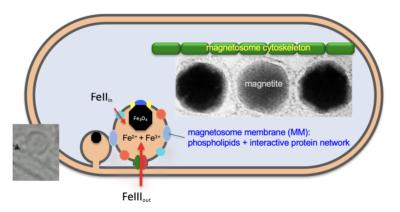
Molekulargenetische Analyse der Magnetosomenbiosynthese
An der Bildung von Magnetosomen sind mehr als 30 Gene beteiligt. Sie bewerkstelligen die Entstehung von intrazellulären Membranvesikeln, die Zielsteuerung von speziellen Proteinen, die Aufnahme von Eisen sowie die Biomineralisation von Magnetitkristallen. In unserer Forschung wollen wir die molekularen Mechanismens dieses komplexen Biosyntheseprozesses aufklären.
Weitere Informationen:
Uebe, R. and D. Schüler. 2016. Magnetosome biogenesis in magnetotactic bacteria. Nature Rev Microbiol, 14:621–637, doi: 10.1038/nrmicro.2016.99.
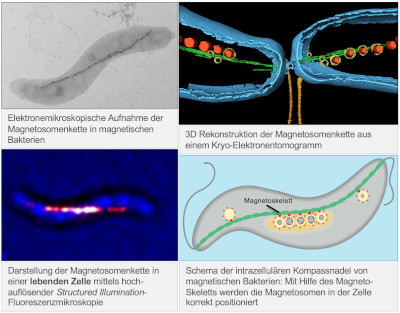
Zellbiologie magnetischer Bakterien
Für ihre optimale Funktion als Magnetfeldsensor werden die einzelnen Magnetosomenkristalle zu einer Kette aufgereiht, die die Zelle - ähnlich einer Kompassnadel - im schwachen Erdmagnetfeld ausrichtet. Die Anordnung und Positionierung der Kette in der Zelle erfolgt entlang eines unerwartet komplexen Netzwerks von verschiedenen Cytoskelett-Proteinen, die das sog. „Magnetoskelett“ bilden, darunter u. a. bakterielle Aktine.
Weitere Informationen:
Müller, F., D. Schüler, and D. Pfeiffer. 2020. A compass to boost navigation - cell biology of bacterial magnetotaxis. Journal of Bacteriology, doi: 10.1128/JB.00398-20.
Toro-Nahuelpan, M., G. Giacomelli, O. Raschdorf, S. Borg, J. M. Plitzko, M. Bramkamp, D. Schüler, and F. D. Müller. 2019. MamY is a membrane-bound protein that aligns magnetosomes and the motility axis of helical magnetotactic bacteria. Nature Microbiol 4, doi: 10.1038/s41564-019-0512-8.
Hintergrundinformationen zur Publikation/Background story - Press release - Presseecho- Cover Nature Microbiology
Biotechnologie: Herstellung, "Engineering" und Funktionalisierung von Magnetnanopartikeln
Neben ihrer biologischen Funktion als zelluläre Magnetfeldsensoren stellen bakterielle Magnetosomen auch magnetische Nanopartikel mit außergewöhnlichen Materialeigenschaften dar. Durch die genetische Kopplung mit verschiedenen Biomolekülen können Magnetosomen mit zusätzlichen nützlichen Funktionen für mögliche Anwendungen in der Grundlagen-forschung und der Biomedizin versehen werden. Weiterhin werden am Lehrstuhl Methoden für die biotechnologische Zucht von Magnet-bakterien und die Produktion von Magnetosomen entwickelt (in Zusammenarbeit mit dem Lehrstuhl f. Bioprozesstechnik). Darüberhinaus haben sich magnetotaktische Bakterien neuerdings als Produzenten von pharmakologisch interessanten Naturstoffen mit cytotoxischer Aktivität erwiesen.
Weitere Informationen:
Mickoleit, F., C. Lanzloth and D. Schüler. 2020. A Versatile Toolkit for Controllable and Highly Selective Multifunctionalization of Bacterial Magnetic Nanoparticles. Small, doi: 10.1002/smll.201906922. Pressemitteilungen
Riese, C.N., R. Uebe, S. Rosenfeldt, A.S. Schenk, V. Jérôme, R. Freitag and D. Schüler. 2020. An automated oxystat fermentation regime for microoxic cultivation of Magnetospirillum gryphiswaldense. Microbial Cell Factories, doi: 10.1186/s12934-020-01469-z.
Awal, R.P.*, P.A. Haack*, C.D. Bader, C.N. Riese, D. Schüler#, and R. Müller#. 2021. Sesbanimide R, a novel cytotoxic polyketide produced by magnetotactic bacteria. mBio, https://doi.org/10.1128/mBio.00591-21. *These authors contributed equally to this work. #corresponding authors.
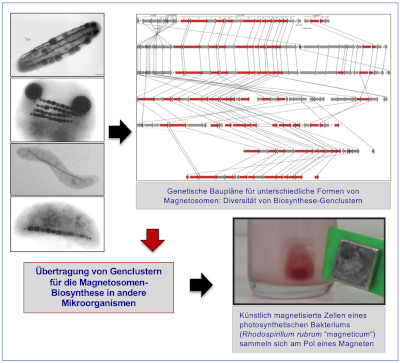
Synthetische Biologie der Magnetosomenbiosynthese
Methoden der modernen Genomanalyse und molekularen Genetik eröffnen die spannende Möglichkeit, die Fähigkeit zur Magnetosomenbildung auch auf andere, natürlicherweise nicht-magnetische Mikroorganismen zu übertragen und dabei „Designer-Magnetosomen“ mit ganz neuen, maßgeschneiderten Materialeigenschaften herzustellen.
Weitere Informationen:
Zwiener, T., M.V. Dziuba, F. Mickoleit, C. Rückert, T. Busche, J. Kalinowski, R. Uebe, and D. Schüler. 2021. Towards a 'chassis' for bacterial magnetosome biosynthesis: Genome streamlining of Magnetospirillum gryphiswaldense by multiple deletions. Microbial Cell Factories, doi: 10.1186/s12934-021-01517-2.
Dziuba, M. V., T. Zwiener, R. Uebe and D. Schüler. 2020. Single-step transfer of biosynthetic operons endows a non-magnetotactic Magnetospirillum strain from wetland with magnetosome biosynthesis. Environmental Microbiology, doi: 10.1111/1462-2920.14950.
Kolinko, I., A. Lohße, S. Borg, O. Raschdorf, C. Jogler, Q. Tu, M. Pósfai, É. Tompa, J. M. Plitzko, A. Brachmann, G. Wanner, R. Müller, Y. Zhang, and D. Schüler. 2014. Biosynthesis of magnetic nanostructures in a foreign organism by transfer of bacterial magnetosome gene clusters. Nature Nanotech. 9:193–197.
Rhodomicrobium als neues Modell für prosthecate multizelluläre Bakterien
Bakterien werden meist als kugel- oder stäbchenförmige Einzeller gesehen, die sich durch Zweiteilung in der Zellmitte vermehren und quasi identische Nachkommen hervorbringen. Eine genauere Betrachtung zeigt jedoch, dass die Evolution eine große Vielfalt an Zellen unterschiedlichster Form geschaffen hat, von denen sich viele asymmetrisch teilen. Außerdem können viele Bakterien Nachkommen hervorbringen, die sich in Form, Funktion und anderen Eigenschaften maßgeblich von ihren Eltern unterscheiden. Auch das Konzept des Einzellers trifft nicht zu, denn Bakterien kooperieren auf vielfältige Weise, und manche bilden sogar mehrzellige Organismen, die aus spezialisierten Zellen bestehen. Über die zugrundeliegenden molekulargenetischen Mechanismen ist oft aber nur wenig bekannt, weil komplexe Bakterien im Labor entweder gar nicht oder nur schwierig zu handhaben sind, oder weil hierfür spezielle Analysemethoden etabliert werden müssen.
Rhodomicrobium kombiniert die oben genannten Eigenschaften auf besondere Weise. Es gehört zu den „knospenden Bakterien“, vermehrt sich also nicht durch Zweiteilung, sondern bildet lange Zellfortsätze (Hyphen) an deren Ende die Nachkommen heranwachsen. Diese können sich wiederum in verschiedene Zelltypen differenzieren. Es ist uns gelungen, das Bakterium für moderne Analysemethoden zugänglich zu machen. Wir möchten unter anderem herausfinden, wie Rhodomicrobium seine langen Zellfortsätze bildet und an deren Ende nicht mehr als vier Nachkommen entstehen lässt. Unbekannt ist auch, wie das replizierte Chromosom seinen Weg durch die langen und dünnen Hyphen in die neuen Zellen findet. Außerdem interessiert uns, wie Rhodomicrobium die Schwelle zur Mehrzelligkeit überwindet und auf welche Weise die Zellen in Verbindung stehen. Hierdurch erwarten wir neue Erkenntnisse über die Entstehung bakterieller Mehrzeller und die zugrundeliegende Steuerung der Zelldifferenzierung.
Weitere Informationen:
Richter, P., B. Melzer, and F.D. Müller. 2023. Interacting bactofilins impact cell shape of the MreB-less multicellular Rhodomicrobium vannielii. PLoS Genetics, doi.org/10.1371/journal.pgen.1010788.
Müller, F.D., Schüler, D., and A. Weig. 2023. Complete genome sequencing and annotation of Rhodomicrobium vannielii strain DSM 166 suggests affiliation to Rhodomicrobium lacus. Microbiology Resource Announcements, doi.org/10.1128/MRA.00690-23.
Dziuba, M.V., F.D. Müller, M. Pósfai, and D. Schüler. 2023. Exploring the host range for genetic transfer of magnetic organelle biosynthesis. Nature Nanotechnology, doi.org/10.1038/s41565-023-01500-5.